Im Skript (S. 11, Abb. 7.2) ist ein Ausschnitt des Hämoglobin-Proteins abgebildet, welches eine Tertiärstruktur darstellt. Bei den meisten Proteinen gibt es keine Quartärstruktur, sondern die Tertiärstruktur entspricht dem ganzen Protein, welches nur in dieser spezifischen 3-D-Form – also die ganze AS-Kette richtig gefaltet – eine gewisse Funktion ausüben kann.
Hämoglobin ist ein Spezialfall: Es gibt insgesamt 4 Untereinheiten (Tertiärstrukturen) bzw. Polypeptidketten, wobei je 2 identisch aufgebaut sind. Jede Untereinheit kann Sauerstoff binden und ist damit funktionell. Diese vier funktionellen Untereinheiten (Tertiärstrukturen) haben sich jedoch zu einer grösseren Struktur, dem ganzen Hämoglobin-Molekül (= Quartärstruktur) vereinigt. In der folgenden Molekülanimation kannst du das nachvollziehen:
- Klicke dich durch die einzelnen Darstellungsoptionen (vgl. oberer Teil des Screenshots).
- Beachte, dass jede Untereinheit eine sauerstoffbindende Nicht-Proteinkomponente trägt, das Häm (im Kalottenmodell dargestellt).
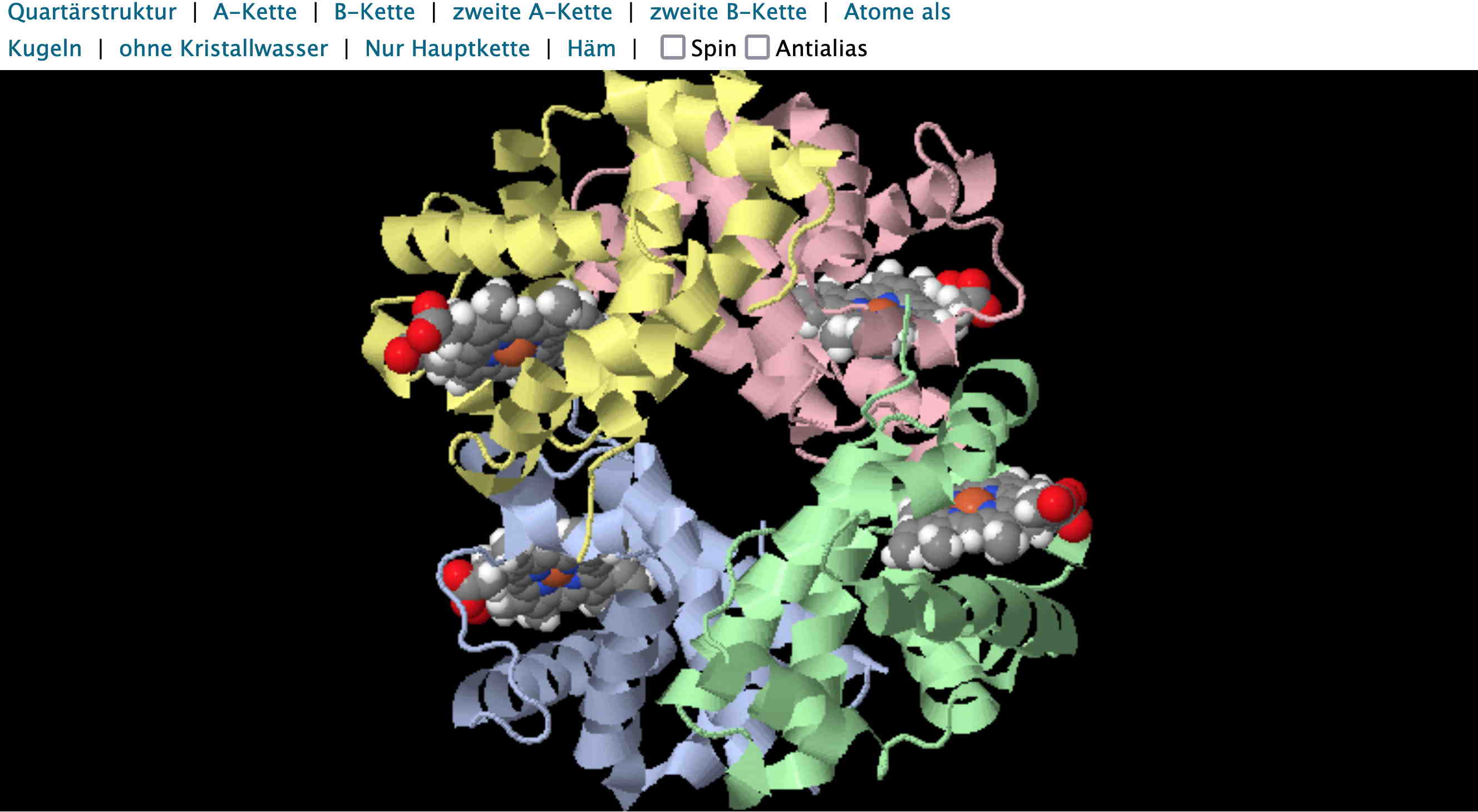
Interaktive Animation (swisseduc) zur Darstellung der vier Untereinheiten (Tertiärstrukturen) des Hämoglobins, welche zusammen die Quartärstruktur, also das ganze Protein Hämoglobin bilden.
Falls du das Hämoglobinmolekül noch selbst in weiteren Darstellungsarten betrachten willst, kannst du mit dieser Animation (Octan) etwas herumpröbeln.