1. Aktives Zentrum eines Enzyms am Beispiel von Pepsin
Der folgende Inhalt basiert auf einer Webseite zu einem Weiterbildungskurs für Mittelschullehrer am biochemischen Institut der Universität Zürich (© A. Honegger). Die auf Java basierenden Jmol-Applets wurden durch Videos und Abbildungen ersetzt. Inhaltlich wurden nur wenige Aspekte ergänzt/präzisiert.
1.1 Pepsin – Beispiel einer sauren Protease
Pepsin ist eine Endopeptidase, die zur Gruppe der Aspartat-Proteasen gehört, da das katalytische Zentrum durch zwei Aspartat-Reste gebildet wird. Mit einem pH-Optimum von ~2 ist diese Peptidase optimal auf die Funktion im sauren Milieu des Magens ausgerichtet. Pepsin spaltet beiderseits von Tyr- und Phe-Resten, langsamer auch saure Aminosäuren und Leucin. Neben Pepsin A gibt es noch das nahe verwandte Pepsin C (Gastricin), das ein pH-Optimum von ~3 und eine etwas engere Substratspezifität zeigt.
1.2 Kalottenmodell von Pepsin
Die Atome sind nach Atomsorten eingefärbt: C = grau, O = rot, N = blau, S = gelb.
Pepsin besteht aus 327 Aminosäuren in einer Kette.
1.3 Hydrophobizität von Pepsin
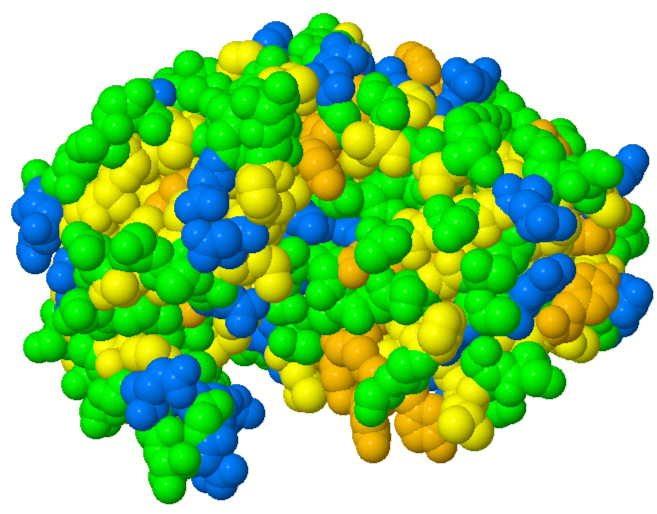
Unpolare, aromatische Aminosäuren (orange): Trp, Phe, Tyr
Unpolare, aliphatische Aminosäuren (gelb): Leu, Ile, Val, Pro, Met, Cys, Ala
Polare Aminosäuren (hellgrün): Ser, Thr, Asn, Gln, (Gly)
Geladene Aminosäuren (blau): Asp, Glu, Arg, Lys, His
1.4 Ladungen von Pepsin bei pH 7
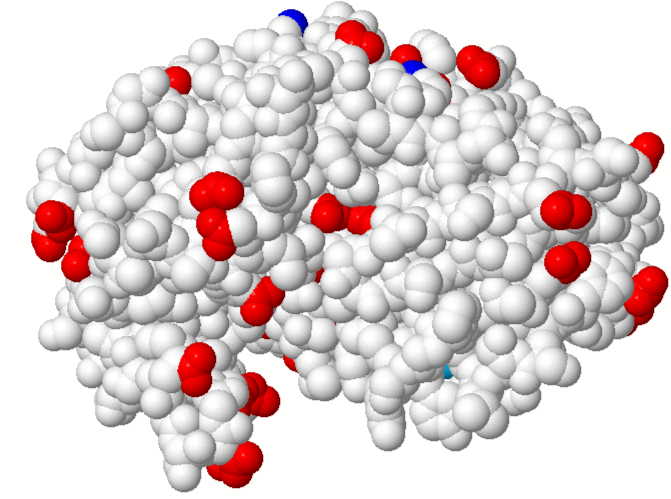
Negativ geladene Reste (rot): Asp, Glu
Positiv geladene Reste (blau): Lys, Arg, His
(Was fällt auf? Wie sähe die Ladungsverteilung im Magen, bei ca. pH 2 aus?)
1.5 Sekundärstruktur von Pepsin
Pepsin enthält hauptsächlich beta-Faltblätter (gelbe Pfeile), aber auch einige kurze alpha-Helices (rote Spiralen) und Beta-Turns (blau). Alle anderen Bereiche stellen die Reste des Proteins (hellgrün) dar.
1.6 Disulfidbrücken von Pepsin
Disulfidbrücken (gelb) zwischen Cysteinresten stabilisieren das Molekül.
1.7 Katalytisches Zentrum von Pepsin
Das aktive Zentrum des Enzyms befindet sich am Grund einer tiefen Spalte, die das Molekül durchzieht. Die katalytischen Aspartat-Reste sind rot (Asp 32) und orange (Asp 215) markiert.
Im Kalottenmodell (vgl. folgendes Video) wird sichtbar, wie eng die Spalte ist, in die das Substrat binden muss. Es verwundert daher nicht, dass denaturierte Proteine deutlich leichter gespalten werden als native Substrate, die mit ihrer kompakten Faltung nicht zur aktiven Stelle gelangen können.
1.8 Mechanismus der Substratspaltung bei Aspartat-Proteasen (Pepsin)
Um die Vorgänge am aktiven Zentrum auf molekularer Ebene zu verstehen, wird am besten der Slider (Playhead) mit der Maus bedient, so dass das Geschehen Schritt um Schritt studiert werden kann.
einklappen